
编者按:我国“十四五”规划提出加快布局DNA存储等前沿数字技术的创新应用。作为遗传信息的载体,DNA本身就是一种天然的优良存储介质。DNA存储了从微生物到人类的亿万生命的遗传信息,并保证生命现象的稳定遗传。近年来,DNA存储在信息存储与处理方面具有的并行性、高存储密度及低能耗等优点,引起来了越来越多科学家的关注。本文主要介绍其相关技术情况。
本期要目
DNA存储及其优点、信道模型的复杂性、应用领域的研究进展以及面临的挑战
主要国家(或地区)DNA存储技术领域规划和举措
DNA存储及其优点、信道模型的复杂性、应用领域的研究进展以及面临的挑战
伴随生命科学的进步,人类在自然中获取灵感。DNA,即脱氧核糖核酸,以其出色的稳定性、高效的可复制性和无出其右的信息密度成为新的储存方式中强而有力的候选。
一、DNA存储及其优点
自然界中,由A, T, C, G这4个核酸碱基组成的脱氧核糖核酸(DNA)承载了所有生物体的遗传信息。DNA存储以脱氧核糖核酸(DNA)生物大分子作为介质,按照一定的编码策略将文本、图片、声音和视频等信息转化为相应的DNA序列,借助生物合成技术合成相应的DNA分子在体内或体外加以贮存,利用DNA分子的特异性杂交技术,如基于聚合酶链式反应(Polymerase Chain Reaction, PCR)[ PCR技术:一种用于放大扩增特定的DNA片段的分子生物学技术,它可看作是生物体外的特殊DNA复制,PCR的最大特点是能将微量的DNA大幅增加。]或磁珠分离技术[ 磁珠分离技术:是一种分子生物学分离技术,它利用其表面修饰的磁性颗粒对生物分子或细胞的亲和结合而进行分离,能对待分离或待检测的靶标进行高效富集,是一种方便、快速、回收率高、选择性强的方法。磁珠分离技术在生物。]访问数据。DNA存储的一般流程如图1所示,主要包括以下6个步骤:
(1)编码:将0,1二进制信息编码为由A, T, C, G组成的DNA序列;(2)合成:利用各种高通量技术合成编码信息的DNA序列;(3)存储:选择合适的载体(体内或体外)将合成的DNA序列进行存储;(4)检索:利用DNA碱基配对的特异性杂交,同特定的引物序列提取DNA分子;(5)测序:对提取到的DNA分子进行测序得到DNA序列;(6)解码:根据解码规则将DNA序列中的信息复原。
相较于传统存储介质,DNA在数据存储方面具有以下4个优点:
(1)存储密度高。DNA可以达到~107GB/mm3,比传统存储介质提高了7个数量级。
(2)保存寿命极长。DNA数据在没有特别人工干预的情况下能保存千年之久。
(3)维护成本极低。DNA数字存储所需要的占地、资源、能源均远远小于传统存储介质。
(4)数据备份十分便捷。PCR为DNA的快速复制扩增提供了技术保障。这些优势有望解决大数据时代电子信息技术对海量数据的有效存储和管理面临的困境。
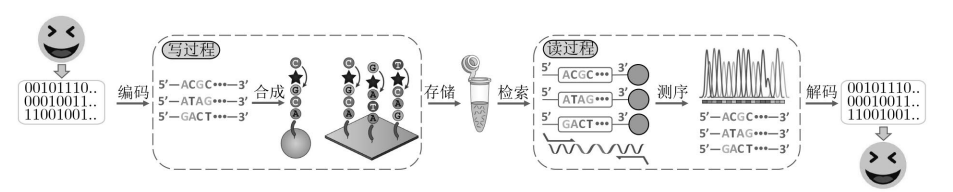
图1 DNA存储的一般流程
二、DNA存储信道模型的复杂性
DNA存储过程主要涉及合成、PCR扩增及测序等技术,可以将DNA存储过程理解为一个信道模型[ 信道模型:在信息论中,信道是指信息传输的通道。在通信中,信道按其物理组成常被分成微波信道、光纤信道、电缆信道等,假定信道的传输特性已知,可以抽象地将信道用模型来描述,并按其输入/输出信号的数学特点以及输入/输出信号之间关系的数学特点进行分类。],该信道模型主要存在由3种技术引起的3种错误。对于一个DNA序列,DNA合成过程将会产生几百到上千个拷贝,DNA合成过程将会发生替换、插入和删除等错误,导致同一序列的每个拷贝将会出现各种不同的3种错误。PCR技术主要用于合成后进一步扩增以及信息读取时对少量样本的扩增,PCR扩增过程也会引入替换错误。测序过程主要会引起替换错误,插入和删除的概率大概在10–6左右。此外,单链DNA分子存储过程也会发生碱基退化变异错误。
丢失是DNA存储过程的另一个重要的问题。在DNA合成过程中,一个序列可能由于各种原因出现合成终止导致丢失。在PCR过程中,由于扩增引物的序列偏好可能导致某些序列出现扩增异常而丢失。测序过程,由于各个序列拷贝分布的不均匀性,可能导致某些序列没有测序导致丢失。
DNA存储信道还存在单链DNA分子分布的不平衡性,主要包括合成过程的不均衡性、PCR扩增过程导致的不均衡性以及测序过程的不均衡性组成。这些不均衡性层层叠加影响,最终导致了测序文件中序列分布的多样性,增加了解码过程的复杂性。
上述错误、丢失及分布复杂性增加了测序文件解码的复杂性。为了解决这些信道“噪声”,常用的解决方案主要包括以下3个方面:
(1)编码设计:基于DNA分子的特性,任何01二进制信息最终翻译的DNA序列应该保证GC含量[ GC含量:在DNA4种碱基中,鸟嘌呤和胞嘧啶所占的比率称为GC含量。]在50%左右,GC含量过大或过小都容易引起合成、PCR及测序过程的错误。引物或地址DNA序列需要有足够大的汉明距离[汉明距离:使用在数据传输差错控制编码里面的,汉明距离是一个概念,它表示两个(相同长度)字对应位不同的数量。]。DNA序列应该尽可能避免产生各种不期望的2级结构。近年,人工合成了另外4种核苷酸,突破性地创造出具有8个字母的DNA分子。碱基数的增加将进一步增加DNA编码的灵活性、鲁棒性[ 鲁棒性:亦称健壮性、稳健性、强壮性,是系统的健壮性,他是在异常和危险情况下系统生存的关键,是指系统在一定(结构、大小)的参数摄动下维持某些性能的特性。]以及编码空间。
(2)物理冗余:主要指通过PCR扩增增加每个DNA序列的拷贝数或者保证序列中的片段重复性,使得同一片段信息出现在不同的DNA序列中。
(3)逻辑冗余:主要指通过各种校验码(如LDPC)或纠错码(如RS码、喷泉码)等,解决各种错误或序列丢失问题。
三、DNA存储应用领域的研究进展
(一)DNA数据库
1995年,基于PCR技术和磁珠分离技术的联想搜索和随机访问的设想被提出。2001年,研究人员设计了一种基于检索链(图2左)和存贮链(图2右)的数据存储方案(如图2)。信息存储链左右两端有一对公共的前向和后向引物序列用于PCR扩增。在前向引物与信息序列之间有一个索引序列用于对信息的检索。检索链主要存储对信息链内容的索引地址。
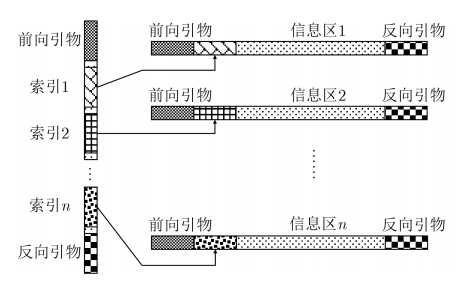
图2 检索链与信息存贮链示意图
2002年,杜克大学的研究团队提出了一种基于数据块的DNA存储结构(如图3所示),每个数据块相当于数据库中的一个字段。当一个数据库由k个块构成,每个块有n个不同的编码信息时,从每个块中取一个特定的编码线性连接后就可以形成一个容量为nk的数据库。他们通过实验构建了一个规模为127的DNA数据库。
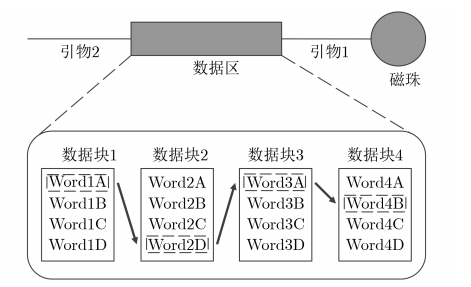
图3 基于磁珠的DNA数据库示意图
显然这种数据库的容量由每个块的编码容量和块的个数决定,大容量数据库需要大量特异性的编码序列集合。DNA计算和DNA存储中的编码问题被系统地阐述,随后一大批学者致力于编码问题的研究。相关团队在模板编码的基础上,提出了模板框的编码理论,可以应用于这种块数据库的编码设计。
一种基于嵌套PCR的存储方式,每个信息的地址由一个块结构构成被提出,信息的检索可以通过其对应的地址块递归做PCR扩增,构建了一个存储容量为16.8M的数据库。2015年,研究人员提出了一种互不相关DNA地址码,构建了一种可重写可随机访问的存储系统。2018年,提出了一种基于关联搜索的图像DNA存储数据库。他们首先将图像用特定个数的元特征表示,然后通过神经网络学习每个元特征图像的编码,使得相似的元特征具有相似的DNA特征编码。
(二)档案文件存储
2012年,研究小组用A/C分别代表奇位/偶位0,T/G分别代表奇位/偶位1,将650 KB数据存入DNA中,使DNA存储数据容量比之前提高了1000倍,从而开启了DNA存储研究的热潮。2013年,首先利用霍夫曼编码将文本或二进制文件转化为三进制的方法被提出。然后,根据当前编码碱基,用其他3个碱基分别代表0, 1, 2转化为DNA编码。此外,为了克服合成及测序错误,采取了如图4(a)所示的4倍信息块冗余编码策略,实现了729 KB的存储总量。2016年,华盛顿大学和微软的研究人员提出了的异或逻辑[ 异或逻辑:异或(xor)是一个数学运算符,应用于逻辑运算。其运算法则为:真异或假的结果为真;假异或真的结果为真;真异或真的结果为假:假异或假的结果为假。]编码策略,成功实现了约151 KB数据的DNA存储。如图4(b)所示,2个信息串A,B异或产生一个新的校验串C(即A⊕B=C),任何1个串的信息可以通过其它2个串得到。这一方法将信息冗余由的4倍降为1.5倍。2017年,纽约基因组中心和哥伦比亚大学计算机系的研究者们提出了DNA喷泉码的编码方法,不仅提高了DNA存储的纠错能力,同时也大大提高了单个碱基的编码位容量(约1.98bit/nt)。他们合成了72000条200nt的DNA序列,总计存储了2.15 MB信息。2017年,结合纠错码和纳米孔测序仪技术,开发了一个便携式的具有随机访问的存储系统。2018年,微软和华盛顿大学的研究小组采用对长序列编码的随机化以及里德-所罗门的纠错策略,用1.34×107条长150bp的DNA链实现了35个文件总计200MB的存储。2019年,天津大学在研究音视频文件的存储中,通过引物池和数据分块机制实现了文件和数据块的随机查找。为了防止各种错误,他们采用采用里德-所罗门(Reed Solomon, RS)码与低密度奇偶校验(Low Density Parity Check, LDPC)码级联编码的纠错方式。苏黎世联邦理工学院提出了一种基于数据块的编码纠错机制,通过内码解决丢失问题,通过外码解决3种错误。以色列理工学院提出了复合DNA码降低编码的冗余性,他们综合了基于二进制序列的喷泉码纠错,以及翻译后DNA序列的RS纠错码。
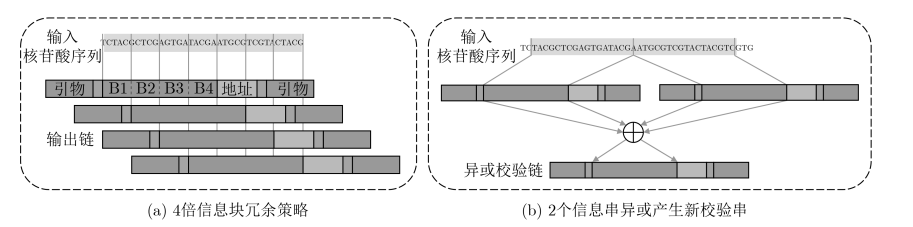
图4 2种数据链冗余设计策略
DNA序列一般由数据区、索引编码区以及两端的扩增引物区组成。DNA存储的“写”(即合成)、“读”(即测序)过程往往容易产生核酸碱基的替换、插入和删除。此外,在数据的存储及检索过程的PCR扩增过程中,也可能产生扩增不平衡导致信息丢失现象。因此,编码技术对于DNA存储系统的稳定性、可靠性及效率至关重要。已有的各种存储研究都采用序列随机化、纠错码以及信息冗余等技术来降低“读”、“写”、“存”过程的错误影响。随着存储容量的增大,构建DNA存储系统所需的码字(codeword)也随之增大。相关团队研究了DNA编码数量的上下限问题,提出了DNA组合码的编码方法降低编码的冗余性。最近,研究团队人工合成了另外4种核苷酸,突破性地创造出具有8个字母的DNA分子。碱基数的增加将进一步增加DNA编码的灵活性、鲁棒性以及编码空间。
(三)活体储存
与体外DNA存储相比,体内存储便于数据的随时复制,但在数据密度上存在一定劣势。此外,活体存储的缺点是生物体中的DNA存在着变异、删除和插入的风险。基于纠错码的变异检测方法被提出。2010年,人工合成了一个支原体基因组(~1.8Mbp)转入酵母细胞,这是将人工信息完整存储在细胞的一次壮举。尽管合成基因组中有4个基因被4 kbp的DNA序列替换,存储于细胞中的DNA仍然可以随着细胞的复制代代相传。2017年,相关研究团队将784Byte和494Byte的4色和21色的图片,以及一部2.6KB的无声短片,通过CRISPR-Cas基因编辑技术存储于细菌活体内,数据还原率达到90%左右。2019年,麻省理工学院的科学家利用基因编辑技术实现了对小分子、光照等生物信号的读/写,该技术可以用来研究细胞的动态响应过程,类似于细胞行为记录仪。
四、DNA存储面临的挑战
首先,从存储的角度,目前合成序列长度一般为100~300碱基,虽然长序列片段的存储效率更高,但是超过此长度的合成成本将急剧增加,特别是合成的费用大约是测序的4个数量级,同时合成和测序的错误率也会随之增加。因此,合成和测序技术尚未适宜大规模工业化应用。
其次,从DNA存储信道模型的角度,编码理论及方法是DNA存储的核心理论问题,高效鲁棒的编码将有望克服目前DNA存储合成、PCR扩增及测序技术的不足,而且可能降低存储的费用。目前的编码基本的流程是将加入纠错冗余信息的二进制字符串直接翻译为DNA序列,这一编码方式将是基于二进制流的纠错,如何结合DNA生化特性,直接研究基于DNA信息流上的组合DNA编码理论将有望解决DNA存储信息道的高可信信息存储问题。
由于DNA存储信息的复杂性,高效可靠的DNA存储需要有坚实的DNA编码理论支撑。目前的DNA存储的编码主要是利用电子信息通讯技术理论建立的各种纠错码的应用。DNA存储需要结合合成、PCR扩增及测序的生化特性,深入研究DNA存储信道的模型及高效的纠错码理论和方法。最后,从信息应用的角度,目前的档案数据存储是一种“死数据”,仍然需要将信息解码为二进制信息由电子计算机做进一步处理分析。基于DNA层次上的随机访问及信息检索技术还处于初步阶段。因此,这一存储模式严重制约了DNA存储在频繁访问大数据领域的应用。(《电子与信息学报》DNA存储及其研究进展)
主要国家(或地区)DNA存储技术领域规划和举措
在全球数据信息总量呈指数级增长的背景下,各国逐渐认识到未来DNA作为存储介质的应用前景以及开发相关新技术的重要性,开始探索DNA存储技术在不同领域应用。高通量DNA合成、测序以及编码作为DNA存储技术三个主要的技术领域,成为各国政策规划布局和技术研发的重点。
一、美国
美国是全球范围内率先对DNA存储技术领域进行研发布局的国家,其多项政策规划均将DNA存储技术领域的相关布局作为一项重要组成部分。2017年3月,美国国防高级研究计划局启动分子信息学计划,旨在发现和明确分子在信息存储和处理中可以发挥的功能,同时为哈佛大学、布朗大学、伊利诺伊大学和华盛顿大学提供约1500万美元的资助,致力于研究和利用各种分子的结构特征和特性来编码和处理数据。同年5月,美国国家科学基金会(National Science Foundation,NSF)发布“针对信息存储和检索技术的半导体合成生物学”项目指南,拨款400万美元用于探索合成生物学与半导体技术之间的协同作用,促进两大领域的新技术突破,增强信息处理和存储能力。2018年7月,美国国家科学基金会公布投入1200万美元资助包括基于DNA的可读取电子存储器、使用嵌合DNA的纳米级芯片存储系统、基于纳米孔读取的高度可扩展随机访问DNA数据存储、核酸内存等在内的8个项目进行研究。美国情报高级研究计划局2018年7月发布了分子信息存储计划,旨在开发可部署的存储技术,减少物理占用空间、功耗和成本。同年10月,在美国国家标准与技术研究院支持下,半导体合成生物学联盟制定第一版《半导体合成生物学路线图2018》,该路线图描述了包含基于DNA的大规模信息存储在内的五个技术领域的技术目标。
2019年以来,美国对DNA存储技术领域仍旧加紧布局。在2019年战略框架报告中,美国国防高级研究计划局在推动科学技术基础研究战略中明确提出重点关注基于分子信息学的新计算方法,并表示将在更广泛的领域去探索除DNA的4个基本分子以外的更多的数据编码处理新方法。2020年2月,美国国家科学基金会发布SemiSynBio-Ⅱ期的项目招标指南,将继续开发与利用结合半导体技术的新兴合成生物学以实现下一代信息存储。美国国防高级研究计划局DARPA小企业项目办公室4月发布SBIR/STTR机会招标合同,邀请提交生物医学技术领域的创新研究概念,拟研发快速、灵活地制造用于合成生物学和治疗应用的DNA分子技术,以能够快速有效地合成高精度千碱基对长度的DNA构建体。此外,佐治亚理工学院、麻省理工学院和哈佛大学、洛斯阿拉莫斯国家实验室、桑迪亚国家实验室和美国陆军研究实验室2020年也获美国情报高级研究计划局资助以进行包括“写入”、测序读取等与DNA存储相关技术的研发。
二、欧盟
欧盟未明确出台与DNA存储相关的政策文件,但对DNA 存储技术领域的规划大多通过未来和新兴技术(Future and Emerging Technologies,FET)欧盟计划下的FET Open进行拨款,资助优瑞卡姆(Eurecom)、法国国家科学研究中心以及DNA合成初创公司海力克斯沃克斯(Helix-works)等开展研究。同时,FET Open下OLI-GOARCHINVE项目聚焦智能DNA存储系统的新技术研究,涉及从编码到测序解码的全领域,将为开发构建智能DNA存储系统所需的基本技术铺平道路。
三、其他国家
除美国和欧洲外,国外其他国家在DNA存储和合成生物学领域也有一定的行动和布局。在合成生物学方面,日本采取了一系列为合成生物学研究人员建立一个共同体的行动,比如2005年成立了日本细胞合成研究协会,其中胚胎科学与技术前期研究为合成生物学项目提供特殊资金等,日本将合成生物学视为其未来科学政策的重要组成部分,并力争在该领域跻身国际前列;2016年日本丰田汽车公司通过“独特的基因样本调整方法”和“下一代基因测序仪”等的成功研究,开发出了快速、低成本DNA解析新技术GRAS,并且与具有丰富DNA解析实绩的日本公益财团上总DNA研究所达成协议,准备对该技术开展进一步的验证评价; 2019年5月由16所合成生物设施机构联合发起的国际合成生物设施联盟(Global Biofoundry Alliance,GBA)在日本神户成立,旨在促进全球合成生物学相关发展等。澳大利亚联邦科学与工业研究组织表示建立包括合成生物学在内的六个未来科学平台,并为之每年投资超过5200万澳元,澳大利亚联邦科学与工业研究组织投资创建的合成生物学未来科学平台(SynBio FSP)旨在支持多领域的创新等来提高澳大利亚的竞争力。
四、中国
我国高度重视DNA存储技术领域的研发,通过对合成生物学等领域专项进行部署和资助。2018年国家重点研发计划合成生物学重点专项共有36个项目,总经费接近7.98亿元。其中专门设置了与DNA存储技术相关的项目。“高通量脱氧核糖核酸(DNA)合成创新技术及仪器研发”项目由中国人民解放军军事科学院军事医学研究院牵头,开发化学法DNA合成新技术、复杂结构序列的高效合成技术和大片段DNA高效组装技术,研制基于高通量芯片的原位组装控制系统及仪器。“使用合成DNA进行数据存储的技术研发”项目由南方科技大学牵头,上海交通大学、中国科学院长春应用化学研究所、福州大学、同济大学联合申报。项目拟开发利用合成DNA高效快速、高密度数据加密编码转码,随机读取,无损解读新方法;开发多类型数据存储DNA介质; 通过合成DNA开发快速编码,存储及数据读取的集成型软件系统。该项目旨在利用新型存储技术应对大数据的爆炸式增长,解决数据快速增长与数据有效存储和利用之间的矛盾,推动我国在DNA数据存储基础研究领域的原始创新和科学突破。2020年,中国科学院深圳先进技术研究院牵头获批7个国家科技部重点研发计划项目,获批“合成生物学”等三个重点专项中总经费8683万元,在“合成生物学”重点专项中,深圳先进院获批4个项目,其中“多方协同合成基因信息安全存取方法研究”项目主要针对DNA存储过程中多方协同操作和安全性问题提出混合加密方法和增量编码技术,进一步探究如何保障合成基因信息多方安全协同与提高 DNA 存储信息高效管理能力,实现合成基因在复杂信息存储需求场景中的存储与可靠读取。(《世界科技研究与发展》DNA存储技术国际发展态势分析)
文件下载:
关联文件: